Dopo un primo intervento, il professor Denis Laloe ha tenuto un secondo interessante seminario sulle basi, anche storiche, della ricerca in campo zootecnico basata sui dati e sulla statistica. E così, oltre a un approfondimento sulla genetica quantitativa, Laloe ha affrontato l'evoluzione storica e le applicazioni moderne delle analisi di integrazione multi-omica con la l’analisi intitolata "Integration of Multiomics Data and Factorial Analysis", presentata ai dottorandi Agrisystem e ai ricercatori del DIANA e del CREI dell’Università Cattolica del Sacro Cuore di Piacenza.
Laloe – ricercatore di fama internazionale specializzato nell’analisi di big data di tipo genetico, zootecnico e ambientale – lavora attualmente all’INRAE (Institut national de la recherche agronomique) di Parigi e collabora con il gruppo guidato dal professor Paolo Ajmone Marsan nell’ambito di un progetto europeo (SCALA MEDI: www.scala-medi.eu) di cui è capofila l’Università Cattolica, con l’obiettivo di migliorare la sostenibilità e la qualità della produzione ovine e avicole sfruttando il potenziale di adattamento delle razze locali del Mediterraneo.
L’approccio data-driven
Partendo dagli anni '60 con l'introduzione degli strumenti di analisi descrittiva da parte di Jean-Paul Benzécri, Laloe ha delineato il percorso che ha portato agli sviluppi contemporanei, tra cui l'analisi fattoriale multipla di Escofier negli anni '80 e l'analisi di co-inerzia di Chessel negli anni '90. Un tema centrale del seminario è stato l'approccio data-driven, che sottolinea l'importanza di lasciare che i dati parlino da soli attraverso un metodo geometrico.
Questo è particolarmente rilevante nell'analisi multiomica, dove i dati sono spesso complessi e, appunto, multidimensionali. Un approccio data-driven garantisce che i modelli riflettano fedelmente le informazioni contenute nei dati, evitando assunzioni preconcette che potrebbero distorcere i risultati.
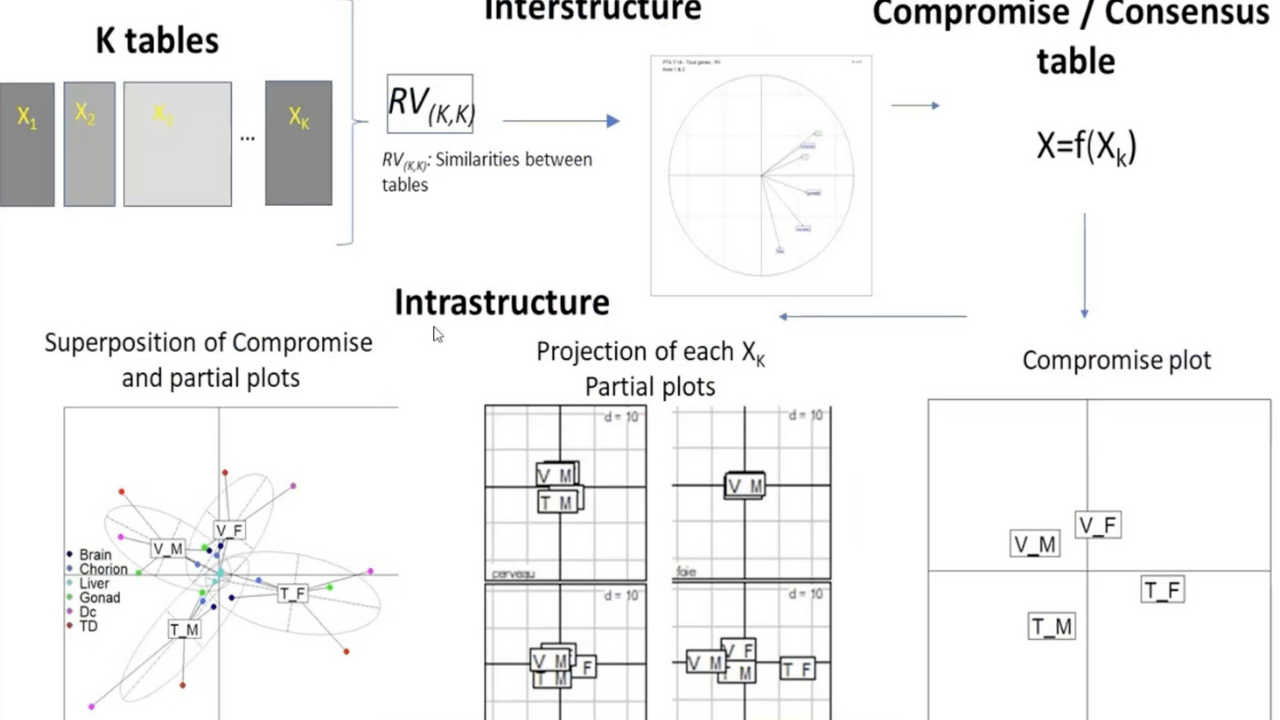
A tal fine, l'approccio geometrico è fondamentale per comprendere le relazioni tra le variabili. In questo contesto, i dati sono rappresentati come nuvole di punti in uno spazio multidimensionale. Questo permette di visualizzare graficamente le relazioni tra le variabili, facilitando l'identificazione di pattern e strutture nei dati. Ad esempio, due variabili possono avere una relazione lineare o non lineare, ma il coefficiente di correlazione può essere lo stesso in entrambi i casi. Tuttavia, la rappresentazione grafica di queste relazioni può rivelare dettagli importanti che il semplice coefficiente di correlazione non può catturare. Un esempio di tecnica geometrica è l'analisi delle componenti principali (PCA). La PCA riduce la dimensionalità dei dati proiettandoli su un numero inferiore di componenti principali che catturano la maggior parte della varianza nei dati. Questo non solo semplifica l'analisi dei dati, ma rende anche più facile visualizzare e interpretare le relazioni tra le variabili. L’ analisi fattoriale multipla (MFA) estende la PCA a situazioni in cui i dati sono strutturati in gruppi di variabili.
Integrazione multiomica
Questo è particolarmente utile per l'integrazione di dati multiomici, dove ogni gruppo di variabili può rappresentare un diverso tipo di dato omico (es. genomica, proteomica, metabolomica). La MFA permette di analizzare simultaneamente questi gruppi, identificando le strutture comuni e le differenze tra di essi. L'integrazione dei dati multiomici è diventata una pratica comune nella ricerca biomedica e nelle scienze della vita. L'integrazione di dati genomici, proteomici e metabolomici permette ai ricercatori di ottenere una visione più completa e integrata dei processi biologici. Ad esempio, i dati genomici possono rivelare variazioni genetiche che influenzano la salute, mentre i dati proteomici e metabolomici possono fornire informazioni sui cambiamenti nei livelli delle proteine e dei metaboliti associati a queste variazioni. Una delle principali sfide dell'integrazione dei dati multiomici è la gestione dei grandi volumi di dati generati. Le tecnologie moderne, come il sequenziamento del DNA ad alta velocità e la spettrometria di massa, producono enormi quantità di dati che devono essere integrati e analizzati in modo efficace. Questo richiede non solo potenti strumenti computazionali, ma anche metodi statistici avanzati in grado di gestire la complessità e l'eterogeneità dei dati. Il continuo sviluppo di nuove metodologie analitiche è essenziale per affrontare le sfide poste dall'integrazione dei dati multiomici. Tecniche come l'apprendimento automatico e l'intelligenza artificiale stanno diventando sempre più importanti per analizzare dati complessi e multidimensionali. Queste tecniche possono aiutare a identificare pattern nascosti e a fare previsioni basate sui dati, migliorando la nostra comprensione dei processi biologici.